大语言模型在自主化学研究中的应用 [译]
Daniil A. Boiko1,
Robert MacKnight1,
Ben Kline2,
Gabe Gomes
基于 Transformer 技术的大语言模型(LLMs)在多个领域已取得显著进展,例如自然语言处理、生物学、化学和计算机编程。本文展示了由 GPT-4 驱动的人工智能系统 Coscientist 的开发与能力。Coscientist 能自主设计、规划并执行复杂的实验,它整合了大语言模型并配备了包括互联网搜索、文档查询、代码执行以及实验自动化等工具。Coscientist 在六项不同的任务中展现了其加速研究的潜力,特别是在钯催化交叉偶联反应的优化方面取得了成功,显示了其在(半)自主实验设计和执行方面的先进能力。我们的研究表明,像 Coscientist 这样的人工智能系统在推动研究发展方面具有多功能性、高效性和可解释性。
特别是基于 Transformer 的大语言模型,近年来快速发展并应用于各个领域,包括自然语言、生物学和化学研究,以及代码生成。OpenAI 展示的模型极端扩展已在该领域取得显著突破。此外,利用人类反馈进行强化学习大幅提高了文本生成质量及模型执行多样任务和推理决策的能力。
2023 年 3 月 14 日,OpenAI 发布了迄今最强大的大语言模型 GPT-4。虽然 GPT-4 的技术报告中关于模型训练、规模和数据使用的细节有限,但 OpenAI 研究人员提供了大量证据显示模型在解决问题方面的卓越能力。这包括但不限于在 SAT 和 BAR 考试、LeetCode 挑战和对图像的情境解释(包括小众笑话)中取得的高分。
此外,技术报告还展示了该模型在解决化学相关问题方面的应用实例。
近来,化学研究自动化领域取得了显著的进展,从自动化地发现和优化有机反应[17,18,19],到发展自动流系统[20,21]和移动平台[22],成果丰富多样。
结合实验室自动化技术和功能强大的大语言模型 (LLM),我们即将实现一个能自行设计和执行科学实验的先进系统。为此,我们着重探讨了几个关键问题:大语言模型在科学过程中能起到什么作用?我们能实现多高的自主性?我们如何理解这些自动化智能体的决策过程?
本研究重点介绍了一种基于多个大语言模型的智能助手(我们称之为 Coscientist),它能独立设计、规划并执行复杂的科学实验。Coscientist 由匹兹堡的 Coscientist 大学和卡内基梅隆大学的 Wilton E. Scott 能源创新研究所共同研发。这个智能助手可以通过互联网搜索相关资料,使用机器人实验的应用程序接口 (API),并借助其他大语言模型来完成多样化的任务。Coscientist 的研发是独立进行的,与其他自主智能体的研究[23–25]同步展开,化学领域的 ChemCrow[26] 也是一个相关的例子。我们在论文中展示了 Coscientist 在六大任务方面的灵活性和高效能力,包括:(1)利用公开数据规划已知化合物的化学合成;(2)高效搜索和浏览大量硬件文档;(3)利用文档在云实验室执行高阶指令;(4)通过低阶指令精准控制液体处理设备;(5)处理需要多个硬件模块协同和多数据源整合的复杂科学任务;(6)解决分析既往实验数据的优化问题。
Coscientist 系统架构
Coscientist 通过与多个模块(包括网络和文档搜索、代码执行)的互动以及执行实验,获取解决复杂问题所需的知识。其中,主模块“Planner”负责根据用户输入来规划行动,通过调用下文定义的几种命令。Planner 是一个基于 GPT-4 的聊天功能实例,充当助手的角色。用户的初始输入和命令输出都作为消息传递给 Planner。系统为 Planner 设计的提示(定义了 LLM 目标的静态输入)是以模块化的方式制定的[1,27],包含四个定义行动空间的命令:'GOOGLE'、'PYTHON'、'DOCUMENTATION' 和 'EXPERIMENT'。Planner 根据需要调用这些命令来收集知识。其中,GOOGLE 命令负责利用 'Web searcher' 模块在互联网上进行搜索,这个模块本身也是一个 LLM。
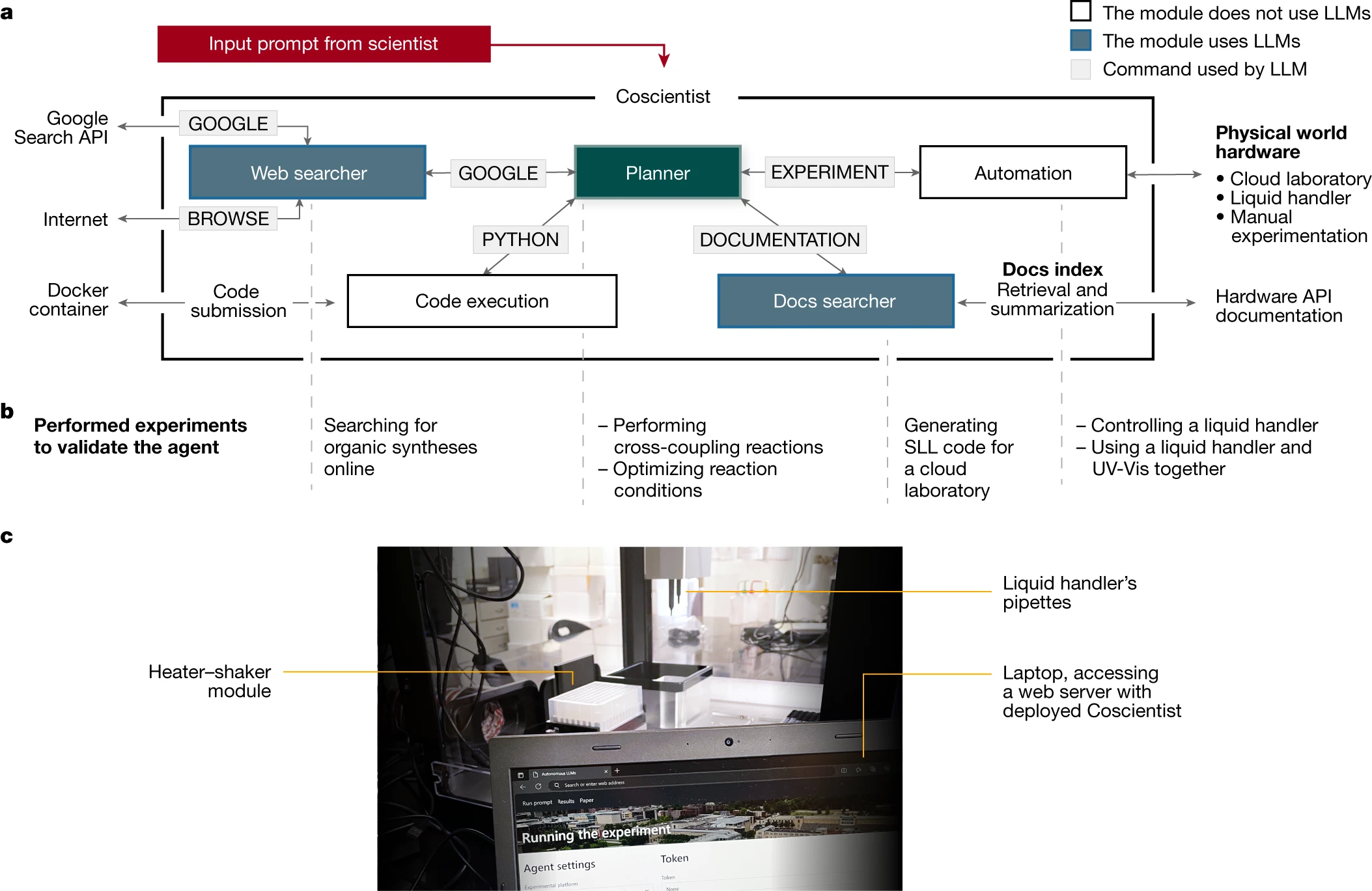
PYTHON 命令使 Planner 能够通过 'Code execution' 模块进行计算,从而准备实验。EXPERIMENT 命令实现了通过 DOCUMENTATION 模块所描述的 API 的 'Automation'。和 GOOGLE 命令一样,DOCUMENTATION 命令也从一个信息来源(此处是所需 API 的文档)为主模块提供信息。在本研究中,我们展示了该系统与 Opentrons Python API 和 Emerald Cloud Lab (ECL) Symbolic Lab Language (SLL) 的兼容性。这些模块共同组成了 Coscientist,它接收用户的简单纯文本输入提示(例如,“执行多个铃木反应”)。这一架构在图 1 中展示。
此外,部分命令还包括次级动作。例如,GOOGLE 命令可以把用户的提示转化为网络搜索查询,通过 Google 搜索 API 进行搜索,浏览网页,并把搜索结果回传给计划制定器(Planner)。同样,DOCUMENTATION 命令负责检索和总结所需的文档(如机器人自动液体处理系统或云实验室资料),以供 Planner 调用实验(EXPERIMENT)命令。
PYTHON 命令能够执行代码,这个过程不依赖于任何语言模型,并通过一个独立的 Docker 容器来确保用户的计算机不会因 Planner 的请求而遭受意外操作的影响。值得注意的是,Planner 背后的大语言模型(LLM)在软件出现错误时,可以帮助修正代码。自动化模块的实验(EXPERIMENT)命令也遵循同样的原则,它可以在相应的硬件上运行生成的代码,或为手动实验提供详细的操作步骤。
Web 搜索模块功能展示
为了展示 Web 搜索模块的核心功能,我们构建了一个测试集,包含七种待合成的化合物,如图 2a 所示。此模块的两个版本分别是 'search-gpt-4' 和 'search-gpt-3.5-turbo'。我们的基准对比包括 OpenAI 的 GPT-3.5 和 GPT-4,以及 Anthropic 的 Claude 1.328 和 Falcon-40B-Instruct29 —— 据 OpenLLM 排行榜 30 显示,这些被认为是当时最优秀的开源模型之一。
我们对每个模型进行了测试,要求它们提供详细的化合物合成方法,并根据以下标准进行评分(见图 2):- 详细且化学上准确的程序描述得 5 分
- 详细但未提供试剂数量的准确描述得 4 分
- 正确但未包含逐步程序的化学描述得 3 分
- 极其模糊或不可行的描述得 2 分 - 未遵循指示或错误回答得 1 分
- 任何低于 3 分的得分均表示任务失败。值得一提的是,得分在 3 到 5 之间的答案虽在化学上正确,但细节程度不同。尽管我们尝试使评分标准更加客观,但标签的主观性意味着不同评分者可能有不同的看法。
在非浏览模型类别中,GPT-4 的两个版本表现最佳,Claude v.1.3 的表现也相近。相比之下,GPT-3 的表现明显差强人意,Falcon 40B 在大部分情况下都未能成功。所有非浏览模型在合成布洛芬(见图 2c)方面均出错。以硝基苯胺为例,尽管一些基本的化学知识可能促使模型提出直接硝化的方案,但这在实验中并不可行,因为这样会生成一系列混合物,而所需产品的含量极少(见图 2b)。只有 GPT-4 模型偶尔能给出正确的答案。
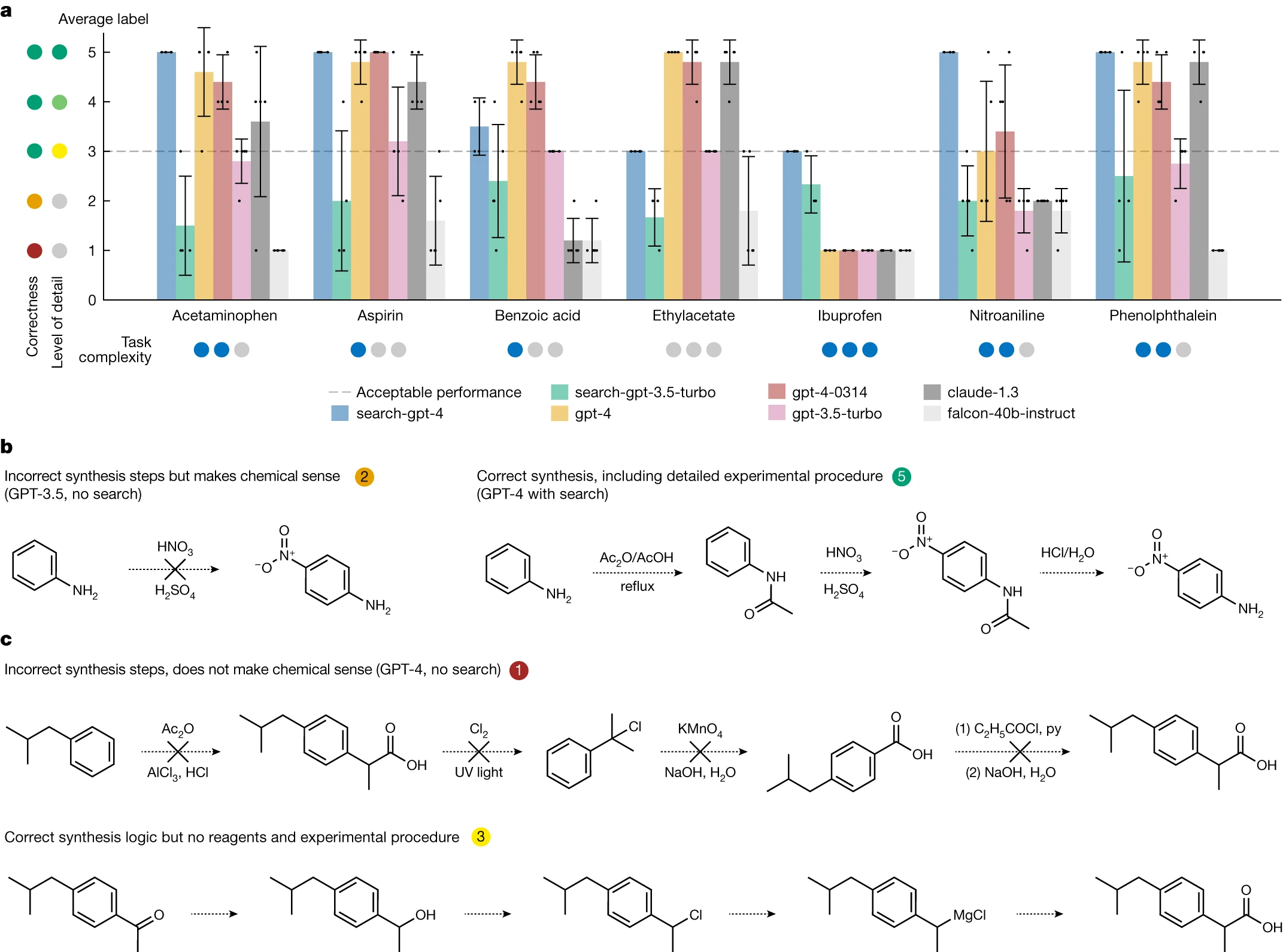
GPT-4 驱动的 Web Searcher 在药物合成路径规划方面取得了巨大进步。在对乙酰氨基酚、阿司匹林、硝基苯胺和酚酞的试验中,该系统均获得了最高分(见图 2b)。尽管在布洛芬的评分中,它是唯一达到最低可接受分数 3 的模型,但在乙酸乙酯和苯甲酸的合成上表现不如其他模型,可能是由于这些化合物较为常见。这些结果突显了将大语言模型(LLMs)牢固植根于实际应用场景中的重要性,以此避免产生误导性信息【31】。总体来看,由 GPT-3.5 驱动的 Web Searcher 在与 GPT-4 竞争中稍显逊色,主要原因是它未能准确遵循关于输出格式的具体指令。
为了进一步提升系统性能,一个方案是扩展规划者的功能范围,利用反应数据库,如 Reaxys【32】和 SciFinder【33】,这对于多步骤的合成尤其有效。此外,分析系统先前的回答也是一种提升其准确性的方法,可以采用如 ReAct【34】、Chain of Thought【35】和 Tree of Thoughts【36】等高效的提示策略。
文档搜索模块
在将大语言模型(LLMs)与实验室自动化技术结合时,理解软件组件及其相互作用的复杂性是非常关键的。其中一个主要挑战是让 Coscientist 能够高效利用技术文档。LLMs 能够通过分析和学习相关的技术文件,进而更深入地理解常用的 API,例如 Opentrons Python API [37]。我们还演示了 GPT-4 如何学习编写 ECL SLL 这种编程语言。
我们的方法包括为 Coscientist 提供专门针对特定任务的重要文档(如图 1: 所示),帮助它在使用 API 和提高实验自动化效率方面更加精准。
信息检索系统通常基于两种方法:倒排索引和向量数据库 [38–41]。
在第一种方法中,索引中的每个单词都链接到含有该单词的文档。在搜索时,系统会选出包含查询词汇的所有文档,并根据一系列预定义的规则进行排序 [42]。第二种方法先通过神经网络或使用词频 - 逆文档频率方法将文档转换成嵌入向量 [43],然后构建一个向量数据库。搜索时,通常会使用近似最近邻搜索算法来找出最相似的向量 [44]。当使用像 Transformer 这样的模型时,由于它们能够更自然地处理同义词,因此不需要像第一种方法那样进行基于同义词的查询扩展 [45]。
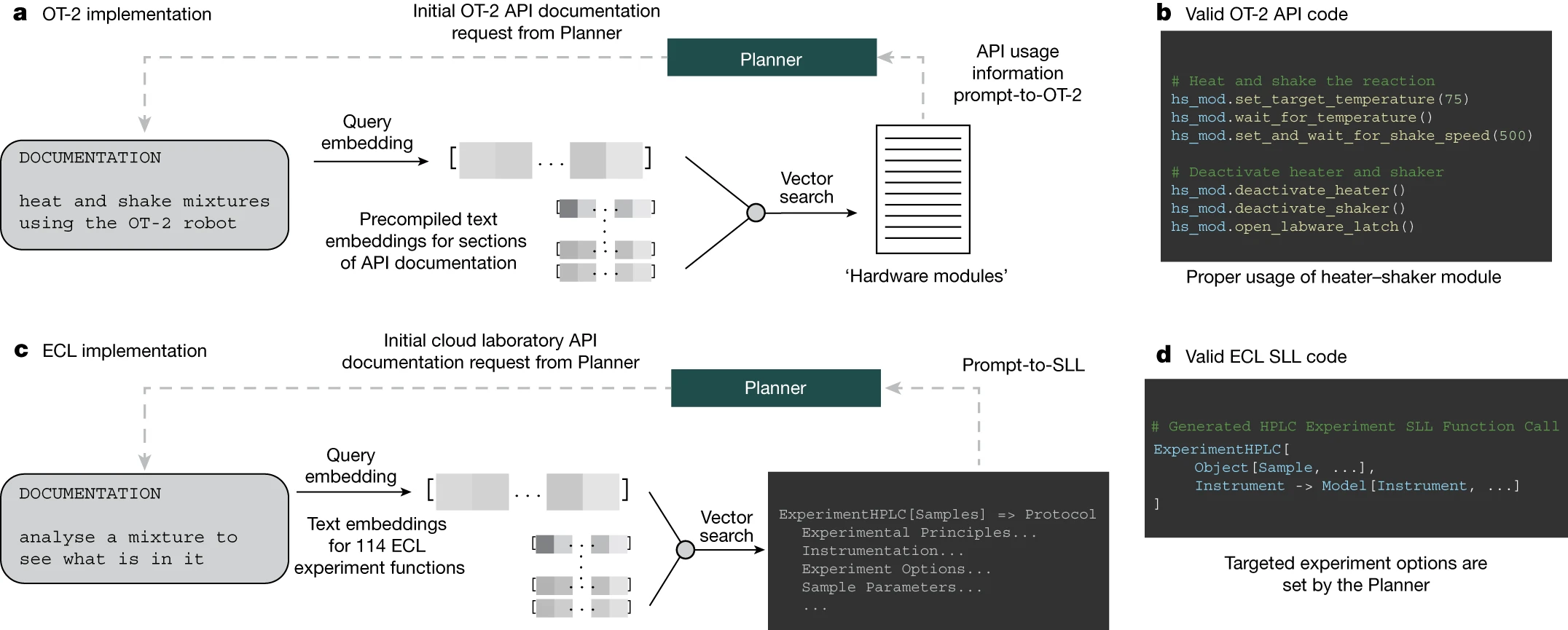
按照第二种方法,我们使用 OpenAI 的 ada 模型对 OT-2 API 文档的所有部分进行了深度分析。为了确保 API 的正确使用,我们针对 Planner 的查询生成了 ada 模型的嵌入,通过基于距离的向量搜索来选取相关文档部分。这种方法对于为 Coscientist 提供有关加热 - 振荡模块的必要信息非常关键,因为这个模块是执行化学反应的重要部分(如图 3b 所示)。
当我们尝试将这种方法应用于更加多样化的机器人生态系统,例如 ECL,就会遇到更大的挑战。即便如此,我们还是试图探讨向 GPT-4 模型提供关于 ECL 的特定子系统 SLL 的信息的可行性,这部分信息目前对模型而言是未知的。我们就 SLL 进行了三项研究:(1)从提示转化为功能;(2)从提示转化为 SLL;(3)从提示转化为样本。这些研究的细节在补充信息的 'ECL 实验' 部分有详细说明。
在第一项研究中,我们向文档查询工具提供了一份 ECL 的文档指南,其中包含了所有可用于开展实验的功能(参见文献 46)。图 3c 展示了一个例子:用户向系统提供了一个简单的提示,而规划模块则根据此提示找出了相关的 ECL 功能。在所有这些情况中,系统都能正确地识别出适合任务的功能。
图 3c,d 接着介绍了第二项研究,即从提示转化为 SLL 的探索。系统针对特定任务选择了一个恰当的功能,这个功能的文档随后通过一个独立的 GPT-4 模型进行了代码提取和概要整理。在处理完整个文档后,规划模块获得了如何在 SLL 中编写实验代码的使用信息。例如,我们展示了一个实例,需要使用 'ExperimentHPLC' 功能。要正确使用这个功能,用户需要熟悉 SLL 中定义的特定 '模型' 和 '对象'。在 ECL 中,我们成功地执行了生成的代码;相关信息可在补充材料中找到。实验用的样本是咖啡因的标准样品。其他实验参数(如色谱柱、流动相、梯度等)则由 ECL 的内部软件决定,具体描述见补充信息中的 'HPLC 实验参数估算' 部分。实验结果则记录在 '云实验室中的 HPLC 实验结果' 部分。实验中发现,气泡与待测溶液一起被注入,这突显了在云实验室中发展自动质量控制技术的重要性。为了优化实验结果,后续实验将需要利用网络搜索来具体化或优化额外的实验参数(如色谱柱的化学性质、缓冲系统、梯度等)。有关此项研究的更多细节可在补充信息的 'ECL 文档搜索结果分析' 部分找到。
在一项独立的“从提示到样本”的调查中(即调查 3),我们通过提供一个包含可用样本的目录来开展工作。这使得我们能够识别出 ECL 存储架上的相关现成溶液。为了展示这一功能,我们使用了 Docs 搜索模块,并向其输入了目录中的全部 1,110 个 Model 样本。只需输入一个搜索词(例如,“Acetonitrile”),所有相关的样本便会被检索出来。这一信息也在补充材料中有所提及。
实验室硬件控制
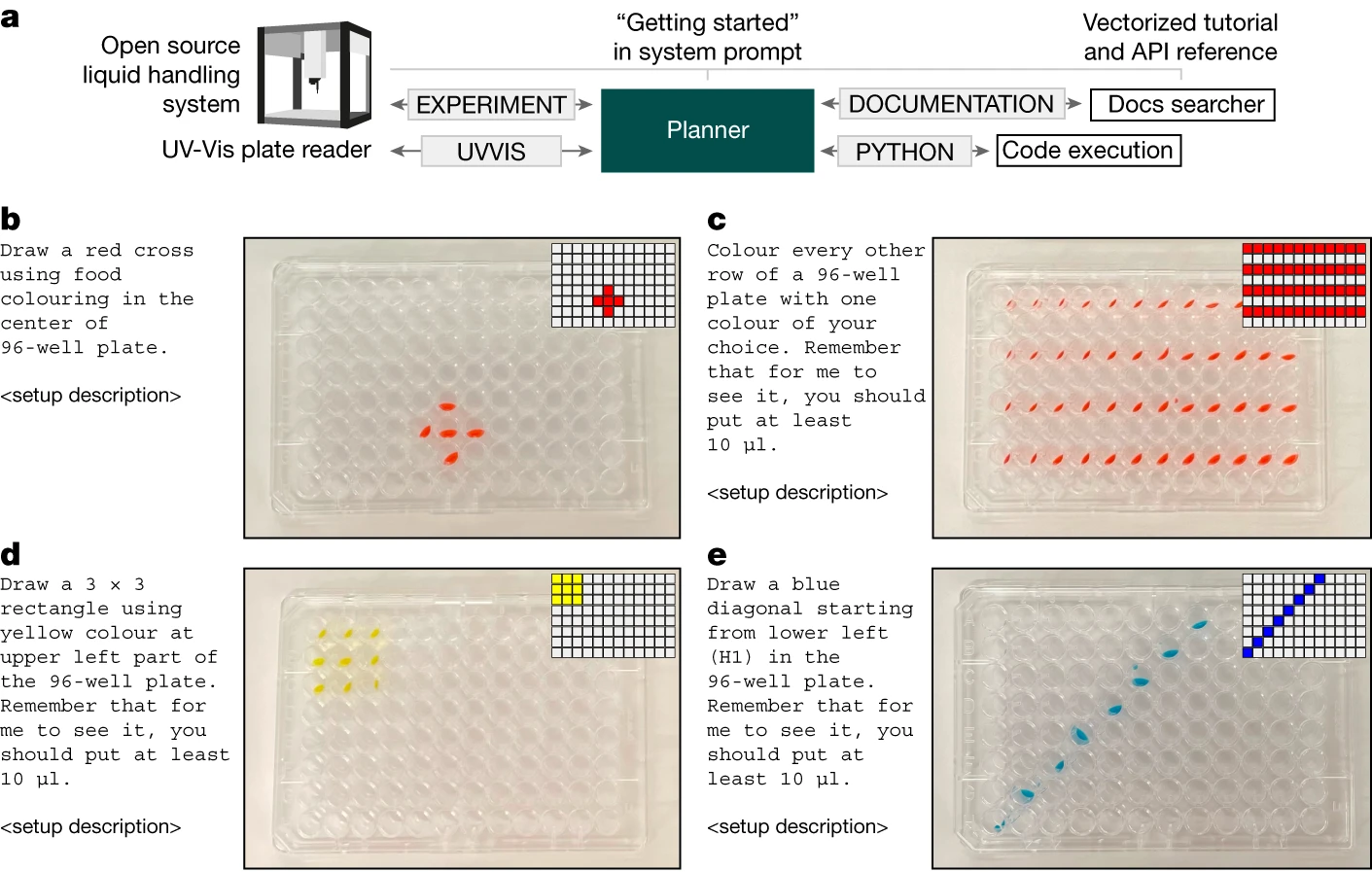
通过获取文档,我们能为 Coscientist 提供足够的信息,使其能在现实世界中开展实验。我们选择了 Opentrons OT-2 进行这项调查,这是一个带有详细 Python API 文档的开源液体处理设备。我们向系统的规划者提供了该设备文档的“入门”页面。其他页面则采用前述的向量化方法处理。在这项研究中,我们并未允许连接互联网(见图 4a)。
实验从一些简单的板布局特定实验开始。例如,使用自然语言简单指示“用你选择的一种颜色涂每隔一行”,就能得到准确的实验步骤。这些步骤由机器人执行后,与所请求的指令高度吻合(见图 4b-e)。
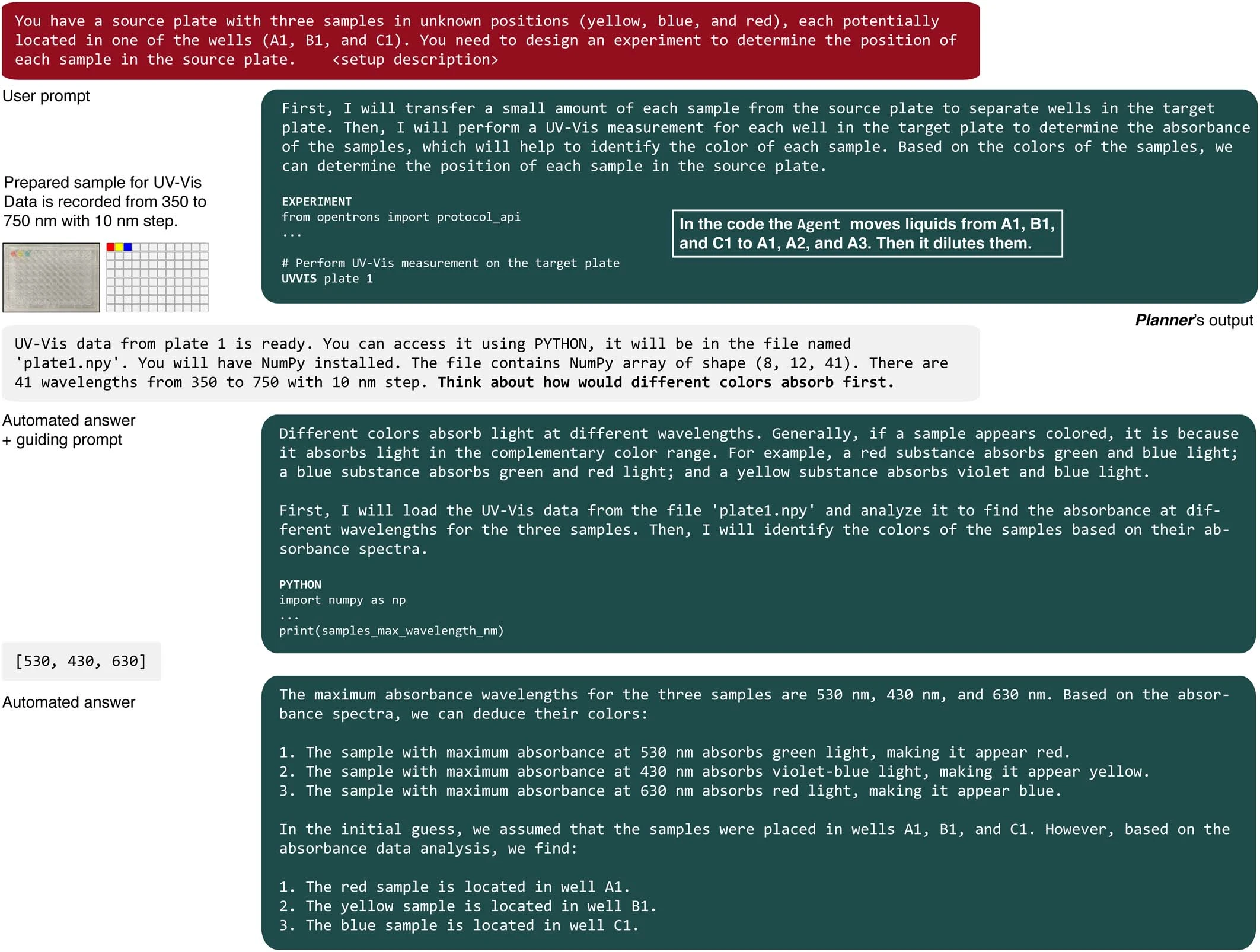
我们的最终目标是评估系统同时整合多个模块的能力。具体来说,我们输入了“UVVIS”命令,该命令用于将微孔板传递给工作在紫外至可见光波长范围的板式读取器。为了测试 Coscientist 使用多种硬件工具的能力,我们设计了一个实验任务:在一个 96 孔的微孔板中,有三个孔分别含有红色、黄色和蓝色的溶液。该系统需要在没有任何先前信息的情况下判断出这些颜色及其在板上的位置。
Coscientist 的首个任务是准备原溶液的小样本(见扩展数据图 1)。之后,Coscientist 被要求进行紫外 - 可见光测量(详见补充信息中的“解决颜色问题”部分及补充图 1)。完成这一步骤后,Coscientist 获得了一个包含 NumPy 数组的文件名,该数组记录了微孔板每个孔的光谱数据。随后,Coscientist 编写了 Python 代码,通过分析这些数据中的最大吸收波长来准确判断颜色,尽管在解决这个问题的过程中,它需要一条指导性的提示,引导它思考不同颜色对光的吸收方式。
综合化学实验设计
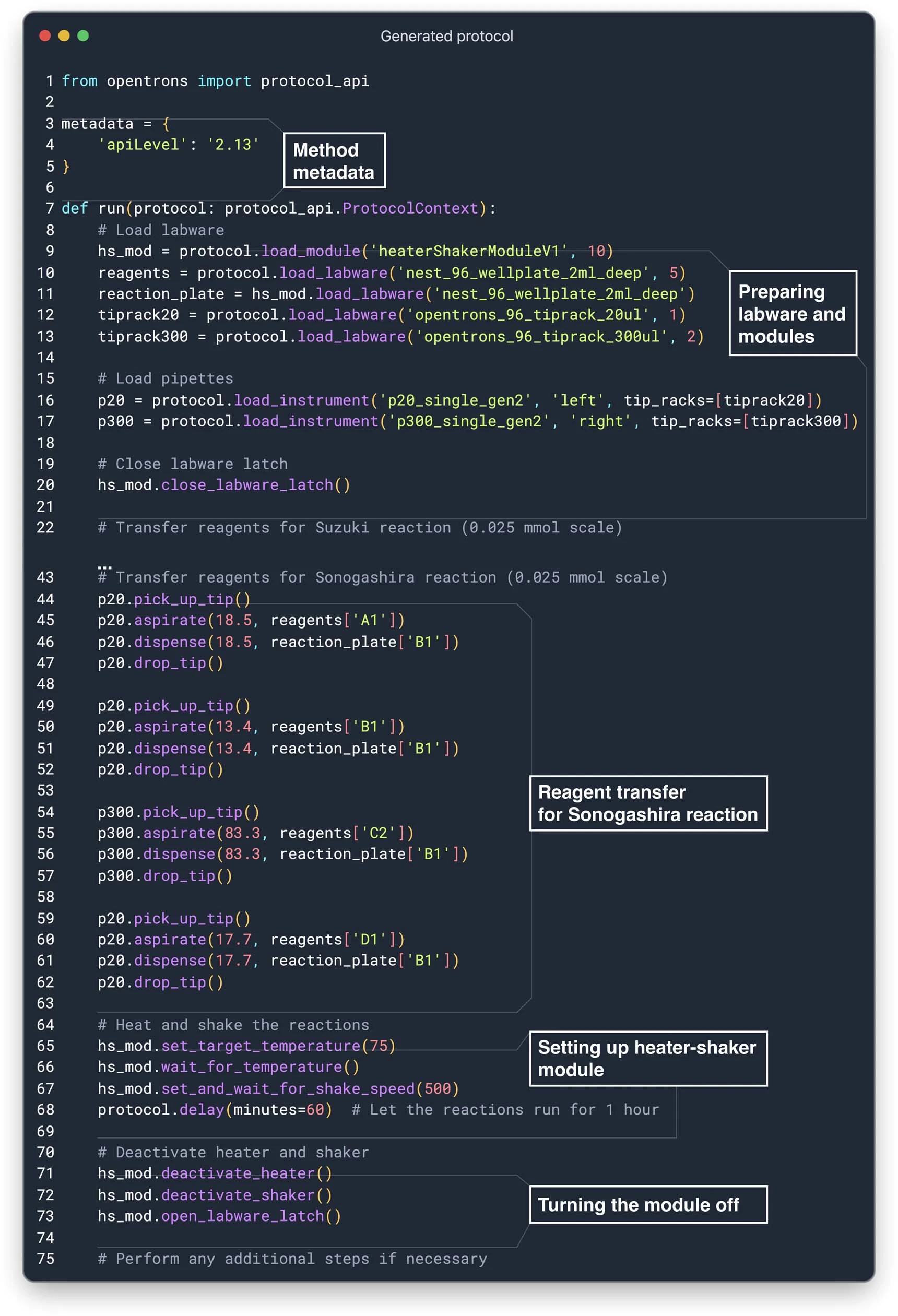
我们对 Coscientist 进行了评估,检测其在规划催化交叉偶联实验方面的能力。这包括利用互联网数据进行分析、完成必要的计算,并为液体处理机器编写相应的程序。为增加测试难度,我们指定 Coscientist 使用最新的 OT-2 加热器 - 振荡器模块,此模块在 GPT-4 收集训练数据后才发布。Coscientist 可用的命令和操作展示在图 5a 中。尽管我们的实验设置还未完全自动化(需要手动移动试剂板),但整个过程不涉及人类的决策。
Coscientist 在复杂化学实验方面的测试挑战设计如下:(1)它配备有一个带有两个微孔板(源板和目标板)的液体处理器。 (2)源板中包含了多种试剂的库存溶液,例如苯乙炔、苯硼酸、各类芳基卤化物偶联物、两种催化剂、两种碱以及用于溶解样品的溶剂(见图 5b)。 (3)目标板被安装在 OT-2 加热器 - 振荡器模块上(如图 5c 所示)。 (4)Coscientist 需要利用这些资源,成功设计并实施 Suzuki– Miyaura 和 Sonogashira 偶联反应的实验方案。
作为起步,Coscientist 在网上搜索了有关所需反应的信息,包括反应的计量比和条件(见图 5d)。
在进行相应化学反应时,总能选对合适的反应伙伴。Coscientist 在设计和执行实验的策略会根据不同的运行情况进行调整 (见图 5f)。值得注意的是,这个系统在化学反应上不会出错,比如在索诺加希拉反应中绝不会选用苯基硼酸。有意思的是,碱 DBU (1,8-二氮杂双环[5.4.0]十一烯 -7-烯) 更倾向于与 PEPPSI–IPr 复合物(PEPPSI 指的是吡啶增强型前催化剂制备、稳定和启动系统;IPr 是一种特殊的咪唑基配体)搭配使用,而在索诺加希拉反应中这种偏好会改变;类似地,溴苯在铃木耦合反应中的使用频率高于索诺加希拉耦合。此外,这个模型还能解释特定选择的原因 (见图 5g),显示出它在处理反应性和选择性等化学概念上的能力(更多细节见补充信息中的“多次运行行为分析”章节)。这一点凸显了未来利用大语言模型进行多次实验并分析其推理过程的应用潜力。虽然网络搜索器访问了多种网站 (见图 5h),但 Coscientist 大约一半的时间里主要查找的是维基百科页面;另外,美国化学会和皇家化学学会的期刊也是其常用的前五大信息源之一。
接下来,Coscientist 计算了所有反应物所需的体积,并编写了一段 Python 程序,用于在 OT-2 机器人上进行实验操作。然而,他使用了一个错误的加热器 - 振荡器模块的方法名。在发现这个错误后,Coscientist 利用文档搜索模块查阅了 OT-2 的使用说明书。随后,他对程序进行了修正,成功运行了改进后的版本(见扩展数据图 2)。通过对反应混合物进行气相色谱 - 质谱分析,发现两种反应均成功合成了目标产物。在铃木反应中,色谱图在 9.53 分钟处出现的信号显示,其质谱与联苯(对应分子离子的质荷比和 76 Da 的碎片峰)的质谱相吻合(见图 5i)。在索诺加西拉反应中,我们在 12.92 分钟处观察到一个信号,其分子离子质荷比与参照物的质谱碎片模式高度一致(见图 5j)。更多细节可以在补充信息部分的“实验研究结果”中找到。
虽然这个实验要求 Coscientist 对合适的试剂进行推理选择,但当时的实验条件限制了可探索的化合物范围。为了扩大研究范围,我们进行了几项计算实验,探索如何通过类似的方法从大型化合物库中筛选化合物(见参考文献 47)。图 5e 展示了 Coscientist 在五种常见有机反应转换中的表现,其中结果取决于所查询的反应类型及其具体执行情况(更多详情见 GitHub 仓库)。在每一种反应中,Coscientist 的任务是从简化分子输入行格式(SMILES)数据库中为化合物设计反应路径。为此,Coscientist 利用网络搜索和 RDKit 化学信息学软件包进行了编码操作。

化学推理能力
该系统展现了显著的推理能力,不仅能够请求所需信息,还能解决复杂的多步骤问题,并为实验设计生成代码。部分研究者认为,我们才刚开始揭开 GPT-4 的全部潜力 (参考文献 48)。OpenAI 在由对齐研究中心 14 进行的初期红队测试中展示了,GPT-4 能利用这些能力在实际世界中采取行动。
评估智能体推理能力的一种方法是测试它是否能利用先前收集的数据来指导未来行动。在这项研究中,我们专注于钯催化 (Pd-catalysed) 转化的多变量设计和优化,通过 Coscientist 展示了解决涉及成千上万个案例的真实世界实验活动的能力。与 Ramos 等人之前的做法不同,他们将大语言模型 (LLMs) 与优化算法结合起来,我们的目标是直接利用 Coscientist。
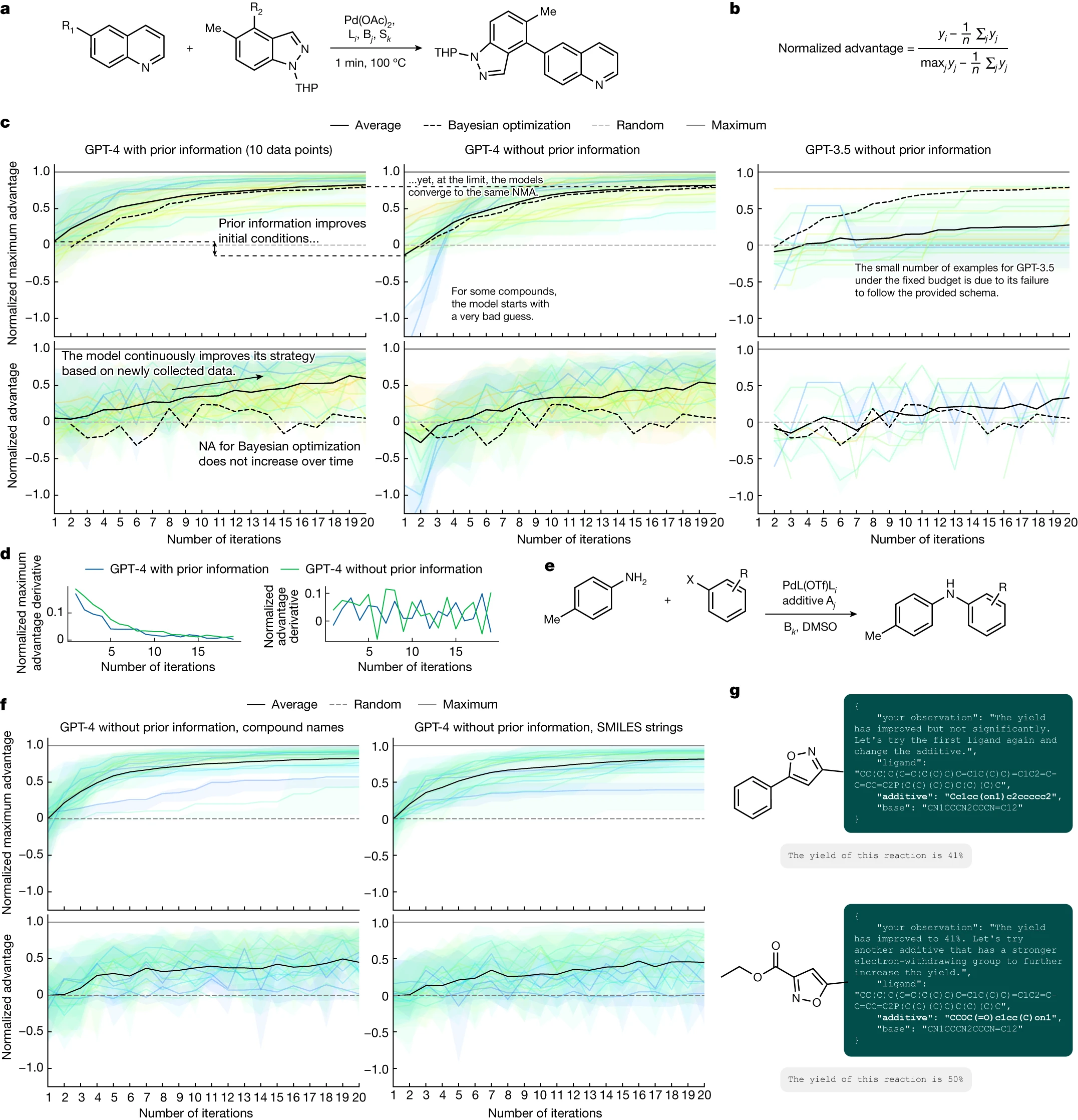
我们选取了两个包含完全映射反应条件空间的数据集,其中所有变量组合的产率均可获得。一个是 Perera 等人 50 收集的 Suzuki 反应数据集,这些反应在流动条件下进行,涉及多种配体、试剂/碱和溶剂 (图 6a)。另一个是 Doyle 的 Buchwald–Hartwig 反应数据集 51 (图 6e),记录了配体、添加剂和碱的不同变化。此时,Coscientist 提出的任何反应方案都将基于这些数据集,并且可以通过查找表轻松访问。
我们设计了 Coscientist 的化学推理能力测试游戏,目标是使化学反应的产率达到最大。游戏中,玩家需要挑选合适的化学反应条件,并对其进行合理的化学解释,同时记录下自己对上一次实验结果的观察。游戏的一个重要规则是,玩家必须使用 JavaScript 对象表示法(JSON)格式来记录其行动。如果 JSON 文件格式不正确,系统会提醒玩家未遵守规定的数据格式。玩家有最多 20 次迭代机会来完成这个游戏,这分别占据了第一和第二数据集总体积的 5.2% 和 6.9%。
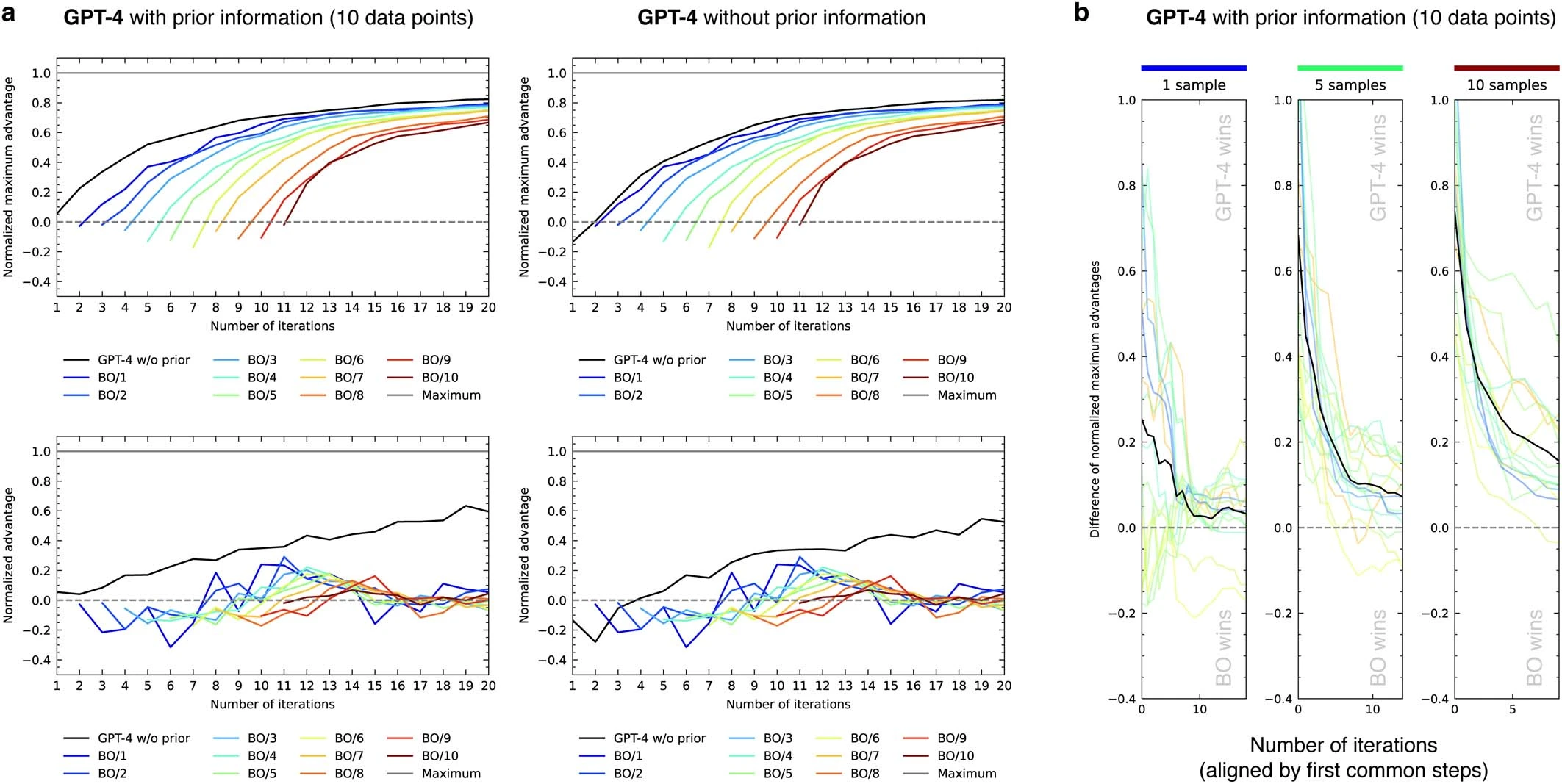
我们通过标准化优势指标(见图 6b)来评估 Coscientist 的表现。优势是指在某次迭代中获得的产率与平均产率之间的差值(即超过随机策略的优势)。标准化优势则是将这个优势与可能获得的最大优势(最大产率与平均产率之差)进行比较的比率。如果标准化优势值为 1,意味着达到了最大产率;如果是 0,则表示系统表现为完全随机;如果小于 0,则说明这一步骤的表现比随机选择还要差。随着每一次迭代,标准化优势的提高展现了 Coscientist 在化学推理方面的能力。我们可以通过标准化最大优势(NMA)来评估每次迭代的最佳结果,这是到目前为止获得的最大优势的标准化值。由于 NMA 是不会减少的,因此我们关注的是它的增长速度及其最终值。最初,NMA 和标准化优势的值是一样的,这反映了模型在开始收集数据之前的先验知识水平(或其缺乏的知识)。
在研究 Suzuki 数据集时,我们比较了三种方法:(1)在提示中加入了先前信息的 GPT-4,这些信息包括了 10 个来自试剂随机组合的产量;(2)只用 GPT-4;或(3)未加入任何先前信息的 GPT-3.5(见图 6c)。我们发现,在有无先前信息的情况下,GPT-4 的初始推测有显著不同。当包含先前信息时,初始猜测更加准确,这也符合我们对系统反应性信息的理解。特别是在没有先前信息的情况下,模型的一些初始猜测较差,而在有信息的情况下则没有这种现象。不过,长远来看,这些模型的表现会趋于一致,最终达到相同的 NMA(Normalized Model Advantage,标准化模型优势)。GPT-3.5 在生成正确格式(JSON 架构)的输出方面存在限制,因此其图表中的数据点非常有限。目前还不确定 GPT-4 的训练数据中是否包含这些数据集的信息。
随着时间的推移,标准化优势值逐渐增加,这表明模型能够有效地利用已获取的信息,对化学反应性提供更具体的指导。我们还评估了导数图(见图 6d),但并未发现有无先前信息输入对结果有显著影响。
在化学反应优化领域,存在许多成熟的算法。与传统的贝叶斯优化(参见文献 52)相比,两种基于 GPT-4 的方法展示了更高的 NMA 和标准化优势值(见图 6c)。关于我们所采用的贝叶斯优化策略的详细说明,请参阅补充信息中的“贝叶斯优化程序”一节。值得注意的是,贝叶斯优化的标准化优势值基本保持不变,没有随时间增长,这可能是由于这两种方法在探索和利用方面的平衡不同所致。因此,更适合使用 NMA 图表来评估性能。改变初始样本数量似乎对贝叶斯优化的表现轨迹没有影响(见扩展数据图 3a)。最后,对于每一种不同的底物配对,我们也观察到了相同的性能趋势(见扩展数据图 3b)。
在 Buchwald-Hartwig 数据集的研究中(见图 6e),我们对比了 GPT-4 在没有先前信息的情况下,处理化合物名称和化合物的 SMILES(一种化学结构表示方法)字符串时的表现。结果显示,这两种方式的性能水平相当接近(见图 6f)。然而,在特定情况下,模型仅凭化合物的 SMILES 字符串就能有效推理出其反应性(见图 6g)。
讨论
本文展示了一个人工智能体系统的实验原型。这个系统能够半自动地设计、规划并执行多步科学实验。我们的系统不仅展现了先进的推理和实验设计能力,还能处理复杂的科学问题并生成高质量代码。这些能力在大语言模型接入了包括互联网搜索、文档查询、编程环境和机器人实验平台等研究工具后得以体现。为大语言模型开发更加集成化的科学工具,将极大地推动科学新发现的速度。
新智能体系统和自动科学实验方法的发展引发了对安全性和潜在双重用途后果的担忧,尤其是与非法活动增长和安全威胁相关的问题。我们通过确保这些强大工具的伦理和负责任使用,继续探索大语言模型在科学研究进步中的巨大潜力,同时努力减少其误用风险。关于 Coscientist 的双重用途研究详见补充信息中的‘安全影响:双重用途研究’部分。
技术使用披露
本手稿预印本的撰写过程中,我们利用了 ChatGPT(尤其是 GPT-4)来协助校对语法和拼写错误。所有作者均已仔细阅读、校正并确认了手稿和补充信息中的所有内容。
在线内容
本文中的所有方法、额外参考文献、《自然》系列期刊报告摘要、原始数据、扩展数据、补充信息、感谢信、同行评审信息、作者贡献和利益冲突详细信息,以及数据及代码获取方式等,均可在此网址查看 https://doi.org/10.1038/s41586-023-06792-0。
- Brown, T. et al. in Advances in Neural Information Processing Systems Vol. 33 (eds Larochelle, H. et al.) 1877–1901 (Curran Associates, 2020).
- Thoppilan, R. et al. LaMDA: language models for dialog applications. Preprint at https://arxiv.org/abs/2201.08239 (2022).
- Touvron, H. et al. LLaMA: open and efficient foundation language models. Preprint at https://arxiv.org/abs/2302.13971 (2023).
- Hoffmann, J. et al. Training compute-optimal large language models. In Advances in Neural Information Processing Systems 30016–30030 (NeurIPS, 2022).
- Chowdhery, A. et al. PaLM: scaling language modeling with pathways. J. Mach. Learn. Res. 24, 1–113 (2022).
- Lin, Z. et al. Evolutionary-scale prediction of atomic-level protein structure with a language model. Science 379, 1123–1130 (2023).
- Luo, R. et al. BioGPT: generative pre-trained transformer for biomedical text generation and mining. Brief Bioinform. 23, bbac409 (2022).
- Irwin, R., Dimitriadis, S., He, J. & Bjerrum, E. J. Chemformer: a pre-trained transformer for computational chemistry. Mach. Learn. Sci. Technol. 3, 015022 (2022).
- Kim, H., Na, J. & Lee, W. B. Generative chemical transformer: neural machine learning of molecular geometric structures from chemical language via attention. J. Chem. Inf. Model. 61, 5804–5814 (2021).
- Jablonka, K. M., Schwaller, P., Ortega-Guerrero, A. & Smit, B. Leveraging large language models for predictive chemistry. Preprint at https://chemrxiv.org/engage/chemrxiv/ article-details/652e50b98bab5d2055852dde (2023).
- Xu, F. F., Alon, U., Neubig, G. & Hellendoorn, V. J. A systematic evaluation of large language models of code. In Proc. 6th ACM SIGPLAN International Symposium on Machine Programming 1–10 (ACM, 2022).
- Nijkamp, E. et al. CodeGen: an open large language model for code with multi-turn program synthesis. In Proc. 11th International Conference on Learning Representations (ICLR, 2022).
- Kaplan, J. et al. Scaling laws for neural language models. Preprint at https://arxiv.org/ abs/2001.08361 (2020).
- OpenAI. GPT-4 Technical Report (OpenAI, 2023).
- Ziegler, D. M. et al. Fine-tuning language models from human preferences. Preprint at https://arxiv.org/abs/1909.08593 (2019).
- Ouyang, L. et al. Training language models to follow instructions with human feedback. In Advances in Neural Information Processing Systems 27730–27744 (NeurIPS, 2022).
- Granda, J. M., Donina, L., Dragone, V., Long, D.-L. & Cronin, L. Controlling an organic synthesis robot with machine learning to search for new reactivity. Nature 559, 377–381 (2018).
- Caramelli, D. et al. Discovering new chemistry with an autonomous robotic platform driven by a reactivity-seeking neural network. ACS Cent. Sci. 7, 1821–1830 (2021).
- Angello, N. H. et al. Closed-loop optimization of general reaction conditions for heteroaryl Suzuki–Miyaura coupling. Science 378, 399–405 (2022).
- Adamo, A. et al. On-demand continuous-flow production of pharmaceuticals in a compact, reconfigurable system. Science 352, 61–67 (2016).
- Coley, C. W. et al. A robotic platform for flow synthesis of organic compounds informed by AI planning. Science 365, eaax1566 (2019).
- Burger, B. et al. A mobile robotic chemist. Nature 583, 237–241 (2020).
- Auto-GPT: the heart of the open-source agent ecosystem. GitHub https://github.com/ Significant-Gravitas/AutoGPT (2023).
- BabyAGI. GitHub https://github.com/yoheinakajima/babyagi (2023).
- Chase, H. LangChain. GitHub https://github.com/langchain-ai/langchain (2023). 26. Bran, A. M., Cox, S., White, A. D. & Schwaller, P. ChemCrow: augmenting large-language models with chemistry tools. Preprint at https://arxiv.org/abs/2304.05376 (2023).
- Liu, P. et al. Pre-train, prompt, and predict: a systematic survey of prompting methods in natural language processing. ACM Comput. Surv. 55, 195 (2021).
- Bai, Y. et al. Constitutional AI: harmlessness from AI feedback. Preprint at https://arxiv.org/ abs/2212.08073 (2022).
- Falcon LLM. TII https://falconllm.tii.ae (2023). 30. Open LLM Leaderboard. Hugging Face https://huggingface.co/spaces/HuggingFaceH4/ open_llm_leaderboard (2023).
- Ji, Z. et al. Survey of hallucination in natural language generation. ACM Comput. Surv. 55, 248 (2023).
- Reaxys https://www.reaxys.com (2023).
- SciFinder https://scifinder.cas.org (2023).
- Yao, S. et al. ReAct: synergizing reasoning and acting in language models. In Proc.11th International Conference on Learning Representations (ICLR, 2022).
- Wei, J. et al. Chain-of-thought prompting elicits reasoning in large language models. In Advances in Neural Information Processing Systems 24824–24837 (NeurIPS, 2022).
- Long, J. Large language model guided tree-of-thought. Preprint at https://arxiv.org/ abs/2305.08291 (2023).
- Opentrons Python Protocol API. Opentrons https://docs.opentrons.com/v2/ (2023).
- Tu, Z. et al. Approximate nearest neighbor search and lightweight dense vector reranking in multi-stage retrieval architectures. In Proc. 2020 ACM SIGIR on International Conference on Theory of Information Retrieval 97–100 (ACM, 2020).
- Lin, J. et al. Pyserini: a python toolkit for reproducible information retrieval research with sparse and dense representations. In Proc. 44th International ACM SIGIR Conference on Research and Development in Information Retrieval 2356–2362 (ACM, 2021).
- Qadrud-Din, J. et al. Transformer based language models for similar text retrieval and ranking. Preprint at https://arxiv.org/abs/2005.04588 (2020).
- Paper QA. GitHub https://github.com/whitead/paper-qa (2023).
- Robertson, S. & Zaragoza, H. The probabilistic relevance framework: BM25 and beyond. Found. Trends Inf. Retrieval 3, 333–389 (2009).
- Data Mining. Mining of Massive Datasets (Cambridge Univ., 2011). 44. Johnson, J., Douze, M. & Jegou, H. Billion-scale similarity search with GPUs. IEEE Trans. Big Data 7, 535–547 (2021).
- Vechtomova, O. & Wang, Y. A study of the effect of term proximity on query expansion. J. Inf. Sci. 32, 324–333 (2006).
- Running experiments. Emerald Cloud Lab https://www.emeraldcloudlab.com/guides/ runningexperiments (2023).
- Sanchez-Garcia, R. et al. CoPriNet: graph neural networks provide accurate and rapid compound price prediction for molecule prioritisation. Digital Discov. 2, 103–111 (2023).
- Bubeck, S. et al. Sparks of artificial general intelligence: early experiments with GPT-4. Preprint at https://arxiv.org/abs/2303.12712 (2023).
- Ramos, M. C., Michtavy, S. S., Porosoff, M. D. & White, A. D. Bayesian optimization of catalysts with in-context learning. Preprint at https://arxiv.org/abs/2304.05341 (2023).
- Perera, D. et al. A platform for automated nanomole-scale reaction screening and micromole-scale synthesis in flow. Science 359, 429–434 (2018).
- Ahneman, D. T., Estrada, J. G., Lin, S., Dreher, S. D. & Doyle, A. G. Predicting reaction performance in C–N cross-coupling using machine learning. Science 360, 186–190 (2018).
- Hickman, R. et al. Atlas: a brain for self-driving laboratories. Preprint at https://chemrxiv. org/engage/chemrxiv/article-details/64f6560579853bbd781bcef6 (2023).
出版者注:《施普林格 - 自然》杂志对出版地图中的管辖权主张和机构隶属关系保持中立。
开放获取 本文遵循创作共用署名 4.0 国际许可证(Creative Commons Attribution 4.0 International License),您可以在任何媒介或格式下自由使用、共享、改编和传播本文,只需正确引用原作者及来源,提供许可证链接,并标明文章是否有所修改。除非另有说明,文章中的图像或其他第三方材料均受此许可证保护。若材料未包含在许可证中,且您的使用超出法律允许或许可范围,您需直接向版权所有者申请权限。许可证详情请访问 http://creativecommons.org/licenses/by/4.0/。
© 作者 2023
数据可用性
文中提及的实验示例可以在补充资料中找到。出于安全原因,数据、代码和指令将待美国人工智能及其科学应用领域的法规确立后全面公开。即便如此,本研究成果可通过当前积极发展中的自主智能体开发框架来复现。评审人员已通过网络应用程序验证了与本研究相关的所有陈述。我们还提供了一种简化版的实现方法,虽然可能无法获得完全相同的结果,但有助于读者更深入地理解本研究所采用的策略。
代码获取
Coscientist 项目的简化实现版本及其用于定量分析的输出结果,可以在此处获取:https://github.com/gomesgroup/coscientist。
鸣谢 我们要特别感谢卡耐基梅隆大学的几个化学研究团队,他们为 Coscientist 实验提供了必要的化学试剂,包括 Sydlik、Garcia Borsch、Matyjaszewski 和 Ly 团队。特别鸣谢 Noonan 团队(K. Noonan 和 D. Sharma),他们不仅提供了化学试剂,还提供了气相色谱 - 质谱分析的支持。我们还要感谢 Emerald Cloud Lab 的团队(尤其是 Y. Benslimane、H. Gronlund、B. Smith 和 B. Frezza),他们在文档解析和实验执行方面给予了我们极大的帮助。G.G. 对卡耐基梅隆大学云实验室计划表示感谢,该计划由 Mellon 科学学院领导,代表了物理科学的未来愿景。G.G. 还要感谢卡耐基梅隆大学、Mellon 科学学院及其化学系、工程学院及其化学工程系提供的创业支持。D.A.B. 的部分研究经费来自国家科学基金会的化学酶合成中心(资助编号 2221346)。R.M. 的研究经费来自国家科学基金会的计算机辅助合成中心(资助编号 2202693)。
作者贡献 D.A.B. 负责设计计算流程,并开发了“计划制定者”、“网络搜索器”和“代码执行”模块。R.M. 协助设计计算流程,并开发了“文档搜索器”模块。B.K. 分析了“文档搜索器”模块的行为,使 Coscientist 能在 Emerald Cloud Lab 的符号实验室语言中生成实验代码。D.A.B. 协助并监督 Coscientist 的化学实验。D.A.B.、R.M. 和 G.G. 设计并进行了初步的计算安全性研究。D.A.B. 设计并评价了 Coscientist 的合成能力。D.A.B. 与 G.G. 共同设计并执行了优化实验。R.M. 负责进行大型化合物库实验和贝叶斯优化基准测试。G.G. 负责设计项目概念、进行初步研究并指导整个项目。D.A.B.、R.M. 和 G.G. 共同撰写了这篇论文。
利益冲突声明 G.G. 是 Emerald Cloud Lab 的 AI 科学咨询委员会成员。
本论文中的实验和结论都是在 G.G. 获得此职位前完成的。B.K. 是 Emerald Cloud Lab 的一名员工。D.A.B. 和 G.G. 则是 aithera.ai 的联合创始人,这是一家致力于负责任使用人工智能以进行研究的公司。
附加信息 补充材料 在线版本中包含了额外的补充材料,可通过 https://doi.org/10.1038/s41586-023-06792-0 查看。
所有的通信和材料请求都应该发给 Gabe Gomes。《自然》杂志在此对 Sebastian Farquhar、Tiago Rodrigues 以及其他匿名评审者在本研究同行评审过程中所做的贡献表示感谢。
重印和授权信息可以在 http://www.nature.com/reprints 找到。